
在生命科学的浩瀚星海中,空间转录组学正像一束穿透迷雾的光,把“细胞在哪里、在做什么、在与谁对话”从想象变成可测量的事实。它让我们第一次有机会把分子表达、组织结构与细胞命运放在同一张坐标系里理解——而这也意味着,真正的挑战不再是“有没有数据”,而是“能不能把数据变成机制、把机制变成主图”。
站在2026新年之际,BioArt携手战略合作伙伴,推出专精系列培训之“Nature 单细胞空间组学1:1复现训练营”。希望用一场真正“硬核、可落地、可交付”的训练,帮助每一位研究者完成从“会跑流程”到“会复现顶刊、会迁移到自己课题”的跃迁:不止讲概念与工具,更以Nature论文为主线,逐面板拆解、源码级精读。
我们相信,未来的空间组学竞争不在“知道使用哪个软件”,而在于谁能更快构建可解释的空间坐标系,谁能更稳建立“位置—互作—信号—表型”的证据链,谁能把结果做成审稿人一眼认可的主图。愿这门课程成为你2026开年的第一份“科研加速器”:让复现不再靠运气,让机制不再停留在猜想,让每一次分析都能沉淀为可复用、可扩展、可发表的能力与作品。
新一年,我们以顶刊为坐标、以源码为桥梁、以主图为目标。欢迎你加入这场空间转录组的“主图复现之旅”,一起把数据变成故事,把故事变成成果。
论文介绍
本次复现的论文如下

https://www.nature.com/articles/s41586-024-08466-x01
课程特色
1.全流程系统教学:
从空间转录组数据结构、QC、注释到出图全流程,配套“保姆级”R/Python代码模板,快速补齐空间数据分析底层能力
2.一对一指导 + 包教包会:
针对你自己的数据(Xenium/VisiumHD/MERFISH等)提供迁移指导:如何选参数、如何可视化空间结构、如何画出和论文一样的图,确保学完能直接上手课题,让你真正学得会、用得上、在大数据分析的加持下做出更有价值的科研成果。
3.AI赋能 + Nature正文拆解:
不是单纯的跑代码,更多的是理解代码背后的生物学意义。当然也不是单纯的只讲思路,而是直接把 Fig.1–Fig.5 的每个 panel 拆到“输入→关键参数→输出→验收标准”,带你做出真正对齐论文的结果
4.源码级系统学习:
从“能跑”到“能改”:带你读懂作者仓库结构、figure pipeline、关键函数与参数选择逻辑,学会把论文代码迁移到自己的项目,而不是“抄完就忘”
5.直播授课 + 录屏回看 + 长期答疑:
直播带跑、课后录屏反复看,资料包(代码/讲义/验收清单/出图模板)全配齐;课程结束答疑不结束,后续自己数据分析过程中遇到问题也是一对一指导答疑,真正做到包教包会,学有所用做出更有价值的研究成果。
02
课程时间
第一期 · 新年开讲
一个月系统教学,实打实包教包会
2026年1月10日周六晚19:00起
每周五、周六、周日晚19:00-22:00
共十三节课
九节课程精讲,四节课答疑和总结
03
课程核心模块
第一节课:AI+Nature文章思路解读:从“图的堆叠”到“机制证据链”
基于Deepseek高效阅读Nature多组学生信文章和评价文章思路的创新性
Deepseek对 论文叙事框架拆解:开篇提出科学问题 → 提出方法框架 → 在小鼠数据证明 → 用互作/信号建立机制 → 用空间扰动给因果 → 人类回译验证
ChatGPT:如何把这篇Nature论文的范式迁移到自己的课题研究中
第二节课:Nature文章中VisiumHD Xenium MERFISH 数据全流程处理(一)
数据读入多种方法和质量控制 QC(细胞层面)
基础可视化与区域裁剪
过滤 + 归一化 + 降维聚类
构建空间邻接图
第三节课:Nature文章中VisiumHD Xenium MERFISH 数据全流程处理(二)
空间统计(组织结构/邻域关系)
邻域富集 nhood_enrichment
共现 co-occurrence
空间自相关 Moran’s I
第四节课:细胞分割
神经网络模型StarDist定位图像中的星形细胞
基于深度学习Cellpose高质量的细胞分割
第五节课:Fig. 1 复现:通过小鼠小肠的空间转录组统刻画CD8 T 细胞在组织空间定位及其转录状态的动态变化。

5.1 将免疫荧光测得的 细胞位置投影到 IMAP 上,按核密度着色展示感染后不同时间点的定位变化。

5.2Xenium 空间转录组数据的多模态结构与对应关系(H&E、IF、DAPI 分割、Leiden 聚类)及局部放大中转录本与标记基因(如 Cd8a/Cd8b/Gzmb/Xist)的空间叠加
5.3 概览处理后的多个时间点 Xenium 数据:联合 MDE 嵌入与原位空间分布,并在局部放大中突出 P14 细胞/簇及 Leiden 聚类随时间的变化
第六节课:Fig. 2 复现:不同空间生态位驱动

6.1 umap图与原位空间图在多个时间点同时展示了小肠组织在空间中的梯度分布

6.2 计算每个基因的 Spearman 相关并以散点图呈现,筛出与空间定位正/负相关的标志基因

6.3 对基因表达做卷积平滑并在各时间点绘制热图,揭示细胞在上皮区室梯度上的转录变化。

6.4 代表性基因(如 Gzma/Gzmb/Itgae/Tcf7) 按表达加权的核密度图,直观展示在不同门控区域的富集位置
第七节课:Fig.3 复现:空间互作网络 + 细胞因子梯度 + CellChat 机制整合

7.1 单细胞分辨率绘制细胞亚群“连接 connectome ,显示不同细胞类型之间的空间相互作用网络。
7.2 Squidpy 相互作用分数网络图(节点为细胞亚群、边为强相互作用),展示不同模块随时间的网络结构变化

7.3 用热图汇总细胞亚群与 P14 细胞 在不同组织生态位的邻域分析

7.4 TGFβ 随数据的表达趋势 变化 ,并用空间图展示其在绒毛中的实际定位分布。

7.5 热图展示了CellChat 计算三个区域的主要“入向信号通路”强度,指出不同生态位驱动的关键信号来源差异。
7.6 热图描绘小肠轴向上的“细胞因子梯度景观”
第八节课:Fig.4 复现:CRISPR扰动验证解析 CXCR3 决定绒毛分布机制

8.1 气泡图比较未感染与感染时间序列中各细胞类型对Cxcl9/Cxcl10的表达水平与表达细胞比例
8.2Cxcl9/Cxcl10在不同时间点的表达趋势曲线,并用空间散点展示其在绒毛中的实际富集位置。
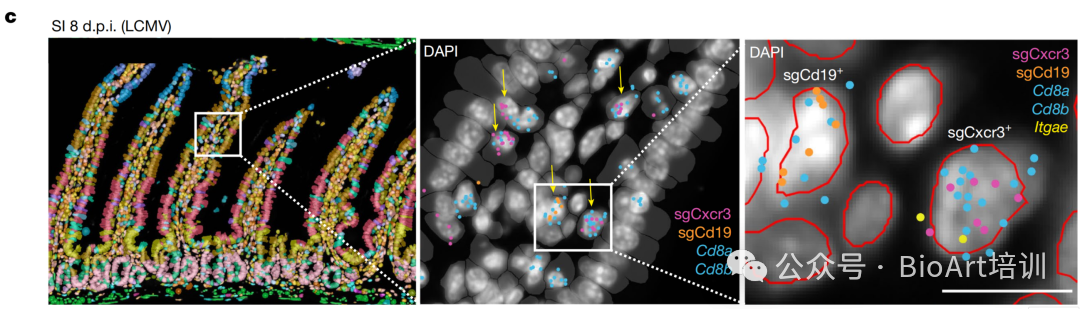
8.3 CD8 T 细胞在组织中的精确位置与细胞核分割及关键转录本信号叠加展示,从而验证基因扰动细胞可在单细胞尺度被空间定位与识别
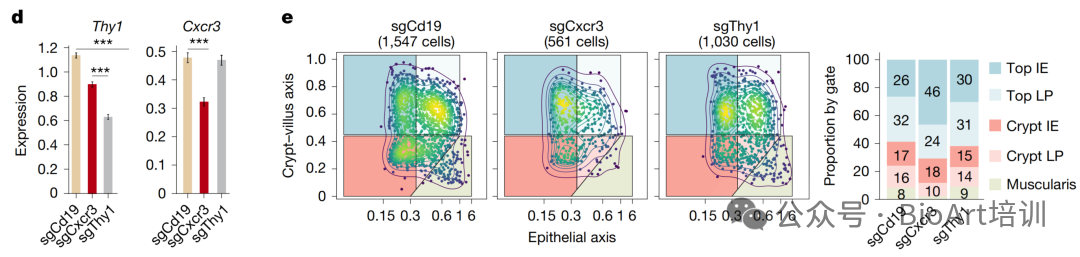
8.4 柱状图展示 目标基因(Thy1、Cxcr3)的表达差异,确认基因扰动有效
8.5 核密度图 量化Cxcr3缺失对定位分布的影响
第九节课:Fig.5 复现:使用人类回肠 Xenium数据进行跨物种验证与细胞互作

9.1 最小失真嵌入图展示不同细胞类型在转录空间中的分离关系。
9.2 比例图展示了各细胞类型在所有切片中的平均相对丰度

9.3 从组织到细胞的多尺度展示:在回肠切片上展示细胞分割与细胞类型/Leiden 聚类,并结合 H&E、DAPI 与转录本叠加。说明 Xenium 数据如何映射到隐窝–绒毛轴与上皮轴。

9.4 核密度展示signature基因集变化
9.5 卷积平滑热图展示 随空间梯度变化的基因表达程序变化

9.6 关键基因表达与不同细胞类型的距离的 Spearman 相关
9.7 CellChat 汇总不同免疫细胞群接收的主要信号通路强度
04
课程费用
课程费用3880元/人,含直播授课+课程录屏反复回看和一对一指导答疑,含 讲义、代码、数据等完整资料包。
团体报名享优惠,请联系培训助手咨询。
七名全职的答疑助理,从早上八点到晚上十二点全天在线 ,365天全年无休的一对一答疑,实打实保证一对一指导的学习效率。
立即扫码,锁定席位


05
合作机构
华哥科研平台
授课理念:将生信内容全部学懂(理解)、学会(会敲代码分析)、学透彻(站在课题顶层设计角度理解)、学以致用(用到自己的标书申请和文章发表中)。
主讲老师
张振华,中山大学博士,目前在东京大学从事医学人工智能研究。深耕单细胞多组学、空间转录组与机器学习领域6年,培养学员3万余人 ; 指导学员发表CNS主刊文章15篇、一区及子刊90余篇 ; 参与国自然重点、国家重大专项、孔雀计划等项目申报;合作院士团队及国际顶尖实验室,发表SCI论文24篇(Mol Cell、PNAS、Cell Rep Med、JACS、Mol Cancer、EMBO Mol Med等顶刊)。
06
课程收获
从“看懂论文”到“复现到一模一样”
将亲手完成Nature 正文 每一个 panel 的 1:1 复现,不是“跑个大概”,而是对齐到坐标范围、统计口径、颜色映射、点大小透明度、图例与标尺等细节。
学完你能做到:看到一张顶刊空间主图,知道它的输入数据是什么、关键步骤在哪里、怎么跑出来、怎么验收对齐。
真正掌握“空间转录组的顶刊核心方法体系”
从形态学或空间几何出发构建可计算坐标系,把组织结构变成可解释的“分区”
KDE 加权的“转录/signature”:把基因/签名投影成空间密度图
卷积表达和空间信号推断
空间互作网络:互作强度→筛边→布局→可视化
空间扰动/因果验证思路:sgRNA 细胞定位→分区比例变化→表达改变→统计检验闭环
让自己的空间数据真正“画得出来、讲得清楚、发得出去”
很多人做空间转录组卡在两件事:图做不出论文质感、结果讲不成机制链条。
这门课会让学员把自己的样本(肿瘤/炎症/感染/治疗前后等)真正用起来:
学会建立组织结构坐标系,把空间异质性从“散点”变成“可解释的梯度”
学会把“空间位置→细胞互作→信号通路→表型变化”的链条做成主图级证据
07
部分代码实战展示































热门跟贴