AI for Chemistry, AI for Drug 领域,各大公司非常关注,英伟达研究团队推出的 ReaSyn 分子合成推理框架,能基于文献中的分子合成数据优化路径设计;谷歌 DeepMind 推出的药物研发专用大模型 TxGemma,可完成生物化学文献筛选、分子结构和作用机制提取、药物特性预测等核心任务。
2025 年,清华大学研究团队发表重磅研究《ReactionSeek: 基于大语言模型的有机合成文献数据挖掘与知识发现》。该研究成功提取了《Organic Syntheses》杂志自 1921 年以来的全部文献数据,自动构建有机合成知识库,而支撑这一成果的核心化学结构式图像识别模块,采用了鹰谷信息的 InDraw 结构式编辑器 AI 图像识别功能。
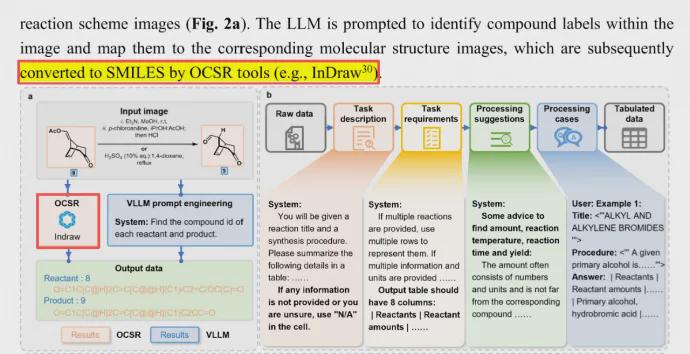
AI for Chemistry,AI for Drug的核心,在于AI能理解化学、生物。在化学领域,AI能理解化学了吗?只有AI能理解化学,才能真正实现AI设计药物分子结构。而AI理解化学的第一步,就是AI结构式图像识别,把化学结构式图片,识别成计算机可处理的化学数据(原理如下图)。

图:InDraw正在进行图像识别
差距几何?国际顶尖化学结构图像识别模型实力拆解
化学结构图像识别是文献数字化的关键环节,直接影响科研效率。当前国际主流顶尖模型以MolScribe与MolNextr为代表,二者凭借创新架构设计与化学知识融合策略,在该领域展现出优异性能,相关技术成果已发表于权威学术期刊,其核心优势与能力均有充分的实验数据支撑,MolScribe与MolNextr的识别能力如下图所示。


来自中国的InDraw AI结构式图像识别,是否和这些国际顶尖化学结构图像识别模型有差距?要客观评判InDraw与MolScribe、MolNextr的识别能力差异,以下借助权威公开测试数据开展量化对比,通过统一的测试标准直观呈现三者的性能表现。
InDraw的AI结构式图像识别实测数据
在CLEF、UOB、JPO、USPTO四大权威公开测试集中,InDraw的识别实力直接“断层领先”同类工具:

UOB测试集拿下99.73%的超高准确率,比MolNextr高出11.23个百分点;CLEF测试集98.62%的成绩,甩开第二名MolNextr足足8.22个百分点;JPO测试集里,InDraw90.22%的表现,比MolNextr领先8.12个百分点;哪怕是竞争最激烈的USPTO测试集,InDraw也以94.67%的准确率,领先MolNextr0.87个百分点,展现了强劲的识别能力!
实际案例
Round 1:模糊图识别对比

在高度模糊化学结构图像的识别任务中,三款模型均展现出较高的识别精度,其中MolScribe仅存在轻微识别误差。从“识别即可用”的实际应用需求来看,仅InDraw可直接满足该标准;MolNextr虽能实现结构的完整正确识别,但分子结构的还原度欠佳,后续需耗费与重新绘制相近的时间用于调整结构位置及角度,难以直接投入实际应用。
Round 2:超大结构识别对比

对于复杂化学结构的识别任务,三款模型的基础识别精度均能满足需求,其性能差异主要体现在结构还原度层面。其中,MolScribe对苯环结构的显示形式局限于凯库勒式,且在结构转换过程中存在轻微苯环扭曲现象,该差异在放大观察时更为显著;MolNextr的识别结果优于MolScribe,可支持苯环的鲍林式显示,但对特殊化学键的还原效果欠佳;InDraw的结构还原度表现优异,基本可实现“识别即用”的需求,此外,MolNextr的识别结果整体亦具备较高质量。
Round 3:2.5D结构识别对比

2.5D结构识别长期以来是化学结构图像识别领域的技术难点。测试结果显示,MolScribe在该类结构识别任务中具备不错的识别精度,但结构还原度表现欠佳,相对位置发生了变化;MolNextr的2.5D结构识别结果实用性较低,基本无法直接投入应用;InDraw能正确识别结构式并保持相对位置,其输出的识别结果可显著降低人工绘制工作量。
小结
实际应用场景中,InDraw的化学结构识别功能表现突出:针对模糊图像、复杂结构及2.5D结构等典型挑战性场景,均能精准捕获核心结构信息,识别结果可较好还原分子真实结构,基本无需人工二次修正。相较之下,同类竞品识别模型的输出结果多需复杂人工校准,其耗时与重新绘制分子结构或无显著差异;而InDraw的识别结果可直接支撑后续科研应用,为您的高效化学探索之旅持续提供可靠支撑。
























热门跟贴